

Secuenciación
Existen diferentes técnicas de secuenciación, pero sin ninguna duda la desarrollada en la década de 1970 por Frederick Sanger, un bioquímico inglés, fue la que permitió la secuenciación a gran escala. Sanger, quien logró la secuencia completa de la molécula de insulina y demostró que las proteínas tienen una estructura específica, desarrolló un método de secuenciación en 1975 (que le valió un segundo Premio Nobel en 1980).
La secuenciación de ADN utilizando el “método didesoxi” de Sanger emplea nucleótidos modificados (didesoxinucleótidos), que no poseen el hidroxilo (OH) en el extremo 3’ . El ADN se sintetiza in vitro utilizando un molde de la cadena que se desee secuenciar, un exceso de sustratos nucleótidos desoxi, una pequeña cantidad de didesoxi específico (A, T, C o G), un cebador o primer y polimerasa.
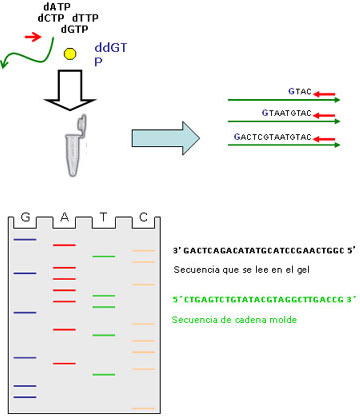
Imagen 1. Secuenciación. Para realizar la secuenciación se coloca en un tubo los cuatro desoxinucleótidos (negro) más un didesoxinulétido (azul), además del primer, el molde y la ADN polimerasa (círculo amarillo). El resultado de la reacción de síntesis son cadenas de distinta longitud. La incorporación de un nucleótido didesoxi hará que termine el proceso de síntesis. Esto se debe a que la polimerasa necesita un grupo hidroxilo en la posición 3’ para poder agregar el siguiente nucleótido (si este grupo no está presente, la polimerasa no puede continuar con la síntesis). Una vez sintetizado el ADN, se realiza una corrida en gel sembrando las productos de las reacciones correspondientes al agregado de cada uno de los nucleótidos didesoxi. De esta manera, se pueden ver distintas bandas correspondientes a tamaños diferentes. Si leemos las calles de abajo arriba (es decir ,de menor a mayor tamaño), tendremos la secuencia del ADN elegido (en orientación 5’ →3’).
Organismos bajo el secuenciador
El primer genoma bacteriano secuenciado fue el de Haemophilus influenzae, el mismo organismo con el que se descubrieron las enzimas de restricción en los años 1960.
Entre los genomas secuenciados figuran los cloroplastos, las mitocondrias de mamíferos, diferentes organismos de laboratorio como levaduras, un gusano nematodo, la mosca de la fruta (Drosophila melanogaster), la planta Arabidopsis thaliana, el ratón Mus musculus. También se han secuenciado una gran variedad de patógenos conocidos como las bacterias causantes del cólera, la tuberculosis, la sífilis, la gonorrea, la enfermedad de Lyme, el virus de la viruela y el del mal de Epstein-Barr.
Con la conclusión de estos y muchos otros proyectos genomas se espera lograr una mejor comprensión sobre el mecanismo de acción de los organismos patógenos, para disminuir su virulencia, o lograr erradicarlos.
 Acerca de educ.ar | Ministerio de Educación de la Nación Argentina
Acerca de educ.ar | Ministerio de Educación de la Nación Argentina ©educ.ar 2006 - Todos los derechos reservados | Privacidad y condiciones de uso | info@educ.gov.ar


